IMPLEMENTAÇÃO COM LIGAÇÃO
Mecanismo básico (fsm)
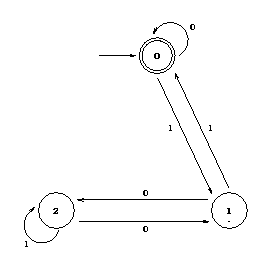
EX:

O autômato do exemplo verifica se uma dada entrada é mútiplo de três.
Nosso objetivo é transformar esse autômato para uma representação em DNA. Como a entrada desse automato trata-se de uma de uma string binária é necessário adequa-la para a representaçao em DNA. Isto é feito com o uso de adaptadores, dupla cadeias de Dna, usualmente com uma cadeia simples como saliência que indica o estado atual além de possuir uma subcadeia que indica o próximo símbolo lido e um loop (cadeia não hibridizada, cujo tamanho depende do números de estados do fsm) que indica o proximo estado .
ALFABETO DE ENTRADA :
simbolo 1 : GAG
simbolo 0 : GCA
O mecanismo de controle é feito por uma molécula dinâmica, essa consiste de uma cadeia dupla de DNA com um segmento que indica o estado atual e outro que indica o último símbolo lido.
ESTADOS :
estado 0 : ttat
estado 1 : gctg
estado 2 : ctca
Molécula inicial : GGGGAGATCttatCTTAA
CCCCTCTAG
O automato do exemplo tera 6 combinações : : 00,01,10.11,20,21
SIMULAÇÃO NO DNA
[0] 0 :
TCt
A t
CCCGGGGAG
atCTTAAGAG
c
tataGAATTGGGCCCCTC
- TAGAATTCTC
[0] 1 :
GCg
A t
CCCGGGGCA
cgCTTAAGAG c
tataGAATTGGGCCCCGT -
GCGAATTCTC
Apenas diferem em GAGATCTTAT para o primeiro e GCATGCGCTG
[1] 0 :
ACc
C t
CCCGGGGAG
caCTTAAGAGc
cgacGAATTGGGCCCCTC -
GTGAATTCTC
[1] 1 :
TCt
A t
CCCGGGGCA
atCTTAAGAGc
cgacGAATTGGGCCCCGT -
TAGAATTCTC
Apenas diferem em GAGCACCTCA para o primeiro e GCAATCTTAT para o segundo.
[2] 0 :
GCg
T c
CCCGGGGAG
tgCTTAAGAGc
gagtGAATTGGGCCCCTC -
ACTAATTCGC
[2] 1:
ACc
C t
CCCGGGGCA
caCTTAAGAG
c
gagtGAATTGGGCCCCTC
- TAGAATTCTC
Apenas diferem em GAGTGCGCTG para o primeiro e GCACACCTCA para o segundo.