Introdução
Biologia molecular é utilizada para indicar uma nova maneira de resolver um problema NP-Completo. A idéia é utilizar tiras de DNA para codificar a instância do problema, e manipular essas tiras usando técnicas comuns disponíveis em qualquer laboratório de Biologia molecular, de forma a simular operações que selecionam a solução do problema, se tal solução existir.
Soluções para problemas NP-Completo usando computação DNA demonstraram que as moléculas de DNA escondem propriedades computacionais interessantes, mas tais resultados não são tão interessantes se um computador molecular não existir. Será que a computação DNA é capaz de realizar qualquer computação ? O projeto de uma máquina de Turing baseada em moléculas de DNA é um passo nesse sentido porque define um sistema computacional molecular capaz de manter um estado e uma memória, e ainda, executar um número indefinido de transições. Vamos descrever duas das propostas feitas de forma a mostrar como computações DNA podem simular o comportamento de uma máquina de Turing.
Modelo de Beaver Para Uma Máquina de Turing Não-Determinística
Para cada passo da computação que vamos descrever, uma molécula de DNA codifica a configuração da máquina de Turing: o conteúdo da fita, o estado atual e a posição da cabeça. Cada mudança de estado requer apenas O(1) operações de laboratório para ser realizada.
Seja M = (Q,![]() ,
,![]() ,
,![]() ) uma máquina de Turing
determinística onde Q é o conjunto de estados (contendo o estado de parada h),
) uma máquina de Turing
determinística onde Q é o conjunto de estados (contendo o estado de parada h), ![]() é um alfabeto finito,
é um alfabeto finito, ![]() é a função de transição de
estados e
é a função de transição de
estados e ![]() é o estado
inicial. Estados e símbolos são codificados usando o alfabeto dos quatro nucleotídeos:
é o estado
inicial. Estados e símbolos são codificados usando o alfabeto dos quatro nucleotídeos: ![]() . Como Q e
. Como Q e ![]() são conjuntos finitos, tal codificação é
obviamente possível e simples. Denotamos com a string
são conjuntos finitos, tal codificação é
obviamente possível e simples. Denotamos com a string ![]() a configuração da máquina de Turing M que está no estado
a configuração da máquina de Turing M que está no estado ![]() , e cuja cabeça de leitura aponta
para o começo da string
, e cuja cabeça de leitura aponta
para o começo da string ![]() e
que tem a string
e
que tem a string ![]() à esquerda
e a string
à esquerda
e a string ![]() à direita. Seja
S(n) o comprimento polinomial da fita M. A configuração de M é codificada por inteiro
em uma tira de DNA simples da seguinte maneira: a presença da sub-tira
à direita. Seja
S(n) o comprimento polinomial da fita M. A configuração de M é codificada por inteiro
em uma tira de DNA simples da seguinte maneira: a presença da sub-tira ![]() indica que o símbolo
indica que o símbolo ![]() está na i-ésima posição da
fita. Da mesma maneira, a presença da sub-tira
está na i-ésima posição da
fita. Da mesma maneira, a presença da sub-tira ![]() indica que M está no estado
indica que M está no estado ![]() e que a cabeça de leitura aponta para a j-ésima posição da
fita. Assim, por exemplo, a configuração
e que a cabeça de leitura aponta para a j-ésima posição da
fita. Assim, por exemplo, a configuração
![]()
é codificada pela tira de DNA
![]()
onde cada ![]() ou
ou ![]() (com
(com ![]() ) é uma sub-tira aleatória oportuna.
) é uma sub-tira aleatória oportuna.
Para realizar uma transição de estado (uma vez que uma tira de DNA simples codifica uma configuração) a idéia é substituir a porção da tira de DNA que é indicada pela transição. Antes de descrever a maneira como uma transição de M é executada, explicaremos a operação biológica que substitui uma porção da tira de DNA.
Vamos supor que temos uma tira M = LAXBR e uma tira Y e queremos
gerar uma nova tira M’ = LAYBR. Obviamente, A, B, L, R, X, Y, ![]() . Para implementar a substituição, operamos da
seguinte maneira: primeiro, as tiras simples
. Para implementar a substituição, operamos da
seguinte maneira: primeiro, as tiras simples ![]() e
e ![]() são
sintetizadas e conectadas com M. O resultado será uma tira parcialmente dupla de
moléculas de DNA onde o lado direito (R) está conectado com o seu complementar, o lado
esquerdo (L) é uma tira simples e a parte central é tal que a molécula
são
sintetizadas e conectadas com M. O resultado será uma tira parcialmente dupla de
moléculas de DNA onde o lado direito (R) está conectado com o seu complementar, o lado
esquerdo (L) é uma tira simples e a parte central é tal que a molécula ![]() está parcialmente limitada com
AXB. Note que, as sub-tiras
está parcialmente limitada com
AXB. Note que, as sub-tiras ![]() e
e ![]() não estão ligadas entre
si ainda, apesar de estarem conectadas com a mesma tira simples M. No próximo passo duas
enzimas são adicionadas: DNA polimerase e DNA ligase. DNA polimerase (usando o fragmento
não estão ligadas entre
si ainda, apesar de estarem conectadas com a mesma tira simples M. No próximo passo duas
enzimas são adicionadas: DNA polimerase e DNA ligase. DNA polimerase (usando o fragmento ![]() como ponto de partida) polimeriza
as moléculas: a tira
como ponto de partida) polimeriza
as moléculas: a tira ![]() é
criada e conectada com M. DNA ligase sela os entalhes, unindo todas as sub-tiras que
estão conectadas com M em uma única tira simples. Esta nova tira simples é exatamente o
complementar de M’, que é a tira que queríamos obter. Esta tira dupla ainda não
está completamente emparelhada com o M original. Neste ponto, a molécula está
desnaturada, deste modo as duas tiras simples são separadas e a molécula L é
adicionada. Desta maneira, a tira simples
é
criada e conectada com M. DNA ligase sela os entalhes, unindo todas as sub-tiras que
estão conectadas com M em uma única tira simples. Esta nova tira simples é exatamente o
complementar de M’, que é a tira que queríamos obter. Esta tira dupla ainda não
está completamente emparelhada com o M original. Neste ponto, a molécula está
desnaturada, deste modo as duas tiras simples são separadas e a molécula L é
adicionada. Desta maneira, a tira simples ![]() se conecta com L. A outra tira simples (a molécula original) é
destruída usando a enzima S1 Nucleasi. A tira que sobrou é polimerizada
começando de L e a molécula M’ é então gerada. O último passo é simplesmente o
desnaturamento da molécula de tira dupla, obtendo a tira simples M’.
se conecta com L. A outra tira simples (a molécula original) é
destruída usando a enzima S1 Nucleasi. A tira que sobrou é polimerizada
começando de L e a molécula M’ é então gerada. O último passo é simplesmente o
desnaturamento da molécula de tira dupla, obtendo a tira simples M’.
Agora podemos mostrar a maneira sugerida por Beaver para realizar a
transição de estado: suponha que a máquina de Turing está no estado q, a cabeça de
leitura está na posição i onde o símbolo ![]() é apontado. Isto é, a codificação da configuração contém a sub-tira
é apontado. Isto é, a codificação da configuração contém a sub-tira
![]()
Além disso, a transição de estado que precisa ser realizada é
![]()
onde ![]() e
e ![]() . A substituição que realiza
esta transição de estado é LAXBR
. A substituição que realiza
esta transição de estado é LAXBR![]() LAYBR, onde
LAYBR, onde
enquanto L e R são respectivamente o lado esquerdo e o lado direito do resto da configuração. Desta maneira uma nova configuração é codificada pela tira de DNA que contém a nova sub-tira
![]()
OBS: A função de codificação E codifica pares de elementos, associando uma posição da fita a um símbolo(ou um estado):
![]()
De modo a fornecer a entrada para a máquina de Turing, um
tubo-teste inicial ![]() precisa
ser criado. Vamos supor que o estado inicial é
precisa
ser criado. Vamos supor que o estado inicial é ![]() e que a fita contém a palavra x como entrada, a
configuração inicial seria
e que a fita contém a palavra x como entrada, a
configuração inicial seria ![]() e sua configuração seria o conteúdo de
e sua configuração seria o conteúdo de ![]() . Além disso, no fim da computação o estado final deve ser reconhecido
(podemos supor que existe apenas um estado de terminação halt) e um resultado (aceita ou
rejeita a entrada) deve ser fornecido. Podemos supor que a entrada foi aceita se e somente
se o símbolo ‘1’ está na primeira posição da fita no fim da computação (e
vice-versa, se o símbolo é ‘0’, então a entrada é rejeitada). Assim, a
operação biológica
. Além disso, no fim da computação o estado final deve ser reconhecido
(podemos supor que existe apenas um estado de terminação halt) e um resultado (aceita ou
rejeita a entrada) deve ser fornecido. Podemos supor que a entrada foi aceita se e somente
se o símbolo ‘1’ está na primeira posição da fita no fim da computação (e
vice-versa, se o símbolo é ‘0’, então a entrada é rejeitada). Assim, a
operação biológica
![]()
realiza a terminação. Ela extrai todas as tiras que contém a sub-tira ![]() , que são as configurações da
máquina de Turing que alcançaram o estado halt e aceitaram a entrada (uma extração
análoga com E(0,1) em vez de E(1,1) extrairia configurações finais onde a entrada é
rejeitada). Se existe pelo menos uma configuração desse tipo (isto é, a operação
detect procura tiras de DNA que foram extraídas), a computação termina e um resultado
oportuno é retornado.
, que são as configurações da
máquina de Turing que alcançaram o estado halt e aceitaram a entrada (uma extração
análoga com E(0,1) em vez de E(1,1) extrairia configurações finais onde a entrada é
rejeitada). Se existe pelo menos uma configuração desse tipo (isto é, a operação
detect procura tiras de DNA que foram extraídas), a computação termina e um resultado
oportuno é retornado.
Finalmente, Beaver sugere expandir este método para simular
máquinas de Turing não-determinísticas: as tiras (que codificam configurações) podem
ser ampliadas a cada passo alcançando um número suficiente para realizar todas as
possíveis transições de estado. Isto é, se o estado atual é q e o símbolo apontado
é ![]() , as tiras são
amplificadas e separadas em
, as tiras são
amplificadas e separadas em ![]() tubos-teste diferentes. Desta maneira, em cada tubo-teste uma transição de estado
oportuna é realizada.
tubos-teste diferentes. Desta maneira, em cada tubo-teste uma transição de estado
oportuna é realizada.
OBS: ![]() é a função de
transição
é a função de
transição
![]()
Uma Implementação Com Restrição Enzimática De Uma Máquina De Turing
Restrições enzimáticas são empregadas na natureza por bactérias para cortar tiras duplas de DNA em sub-tiras específicas chamadas sítios de restrição. Muitas delas cortam DNA em dois pedaços deixando sticky ends (finais pegajosos) nas duas novas tiras. Um exemplo é a enzima EcoRI que corta DNA da seguinte maneira:
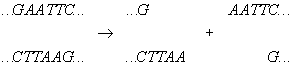
O sítio de restrição é o que está à esquerda da flecha e o produto da restrição são as duas sub-tiras mostradas à direita. Elas têm sticky ends no sentido que elas permanecem parcialmente simples na extremidade e podem ligar-se com um outro sticky end complementar. A enzima EcoRI pertence à classe II Endonucleases. Uma das propriedades desse tipo de enzima é que as duas tiras simples que compõem o sítio de restrição são idênticas: como cada tira de DNA tem uma polaridade, e como duas tiras simples complementares são anti-paralelas.
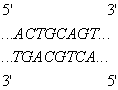
Se lermos, por exemplo, o sítio de restrição da enzima EcoRI (acima) do final 5’ para o final 3’ (ou vice-versa), podemos ver que elas são a mesma tira: ACTGCAGT (palíndromos). Consequentemente, os dois sticky ends gerados pelo corte também são idênticos. Esta simetria permite que um sticky end de uma tira de DNA cortado por uma enzima possa se conectar com um sticky end de outra tira de DNA que tenha sido cortada pela mesma enzima. Esta propriedade da classe de enzimas II Endonucleases é a base da idéia de Rothemund para implementar uma máquina de Turing.
Em poucas palavras, qualquer descrição instantânea de uma máquina de Turing é codificada em uma tira de DNA e a transição de estados é realizada cortando-se a tira em uma sub-tira oportuna (reconhecida pela enzima associada com a transição) e inserindo uma nova sub-tira (oportuna) entre os dois sticky ends para obter a nova descrição instantânea.
Para ser mais preciso, os elementos da máquina de Turing são classificados em constantes e variáveis. A tabela de transição é constante, enquanto a descrição instantânea (que chamaremos de ID) é variável. Cada ID é codificado em uma molécula de DNA simples e circular, chamada plasmid. A tabela de transição de estados (que é a informação constante) é codificada permanentemente pelos oligonucleotídeos. Estas tiras são oportunamente escolhidas e cada uma delas realiza (com poucos passos químicos) a transição correspondente e transforma o plasmid em um novo plasmid que representa o novo ID. Isto é obtido da seguinte maneira: o plasmid codifica os símbolos da fita concatenando fragmentos duplos que codificam cada elemento. O estado atual e a posição da cabeça de leitura também são codificados no plasmid. Ambos aparecem perto da codificação do símbolo apontado pela cabeça de leitura e ambos consistem de subseqüências que contém um sítio de restrição. Cada estado tem o seu sítio e, consequentemente, sua enzima. O sítio e a enzima também dependem do símbolo que a cabeça de leitura está lendo: assim, existe um sítio diferente para cada entrada na tabela de transição. A tabela é codificada por um conjunto de tiras duplas, uma para cada transição da máquina de Turing. Cada um desses oligonucleotídeos contém as codificações do novo símbolo, o do novo estado e o do movimento (para a esquerda ou para a direita) que a máquina de Turing deve realizar para fazer a transição de estado associada com aquele oligonucleotídeo. Finalmente, o oligonucleotídeo tem dois sticky ends que permitem que ele se insira exatamente no sítio que foi cortado pela enzima associado com o estado anterior, realizando assim a transição de estado. Esta substituição do fragmento do plasmid resulta em um novo plasmid que codifica a descrição instantânea desejada, incluindo a codificação do novo estado e a nova posição da cabeça de leitura. Esta operação é realizada com poucas operações químicas que, primeiro cortam o plasmid anterior e desprende o fragmento que deve ser removido, e então circulariza a molécula de DNA com DNA ligase depois que o novo fragmento é inserido.
Existe uma diferença entre esta idéia e aquela proposta por Beaver porque aqui a codificação dos novos elementos já está nas moléculas que codificam a tabela de transição (que Beaver não codificava) e estes elementos não precisam ser sintetizados a cada transição. No método de Rothemund, a tabela é codificada uma vez no começo da computação e cada entrada é associada com enzimas de restrição comercialmente disponíveis.
Rothemund projetou a implementação da máquina de Turing BB-3, que é uma máquina com 3 estados e um alfabeto de 2 símbolos. Já que o produto destes dois números é 6, apenas 6 enzimas diferentes são necessárias porque este é o número de entradas da tabela de transição. Rothemund afirma que sua implementação de uma máquina de Turing é possível para máquinas onde este produto chega até no máximo 60, já que esse é o número de enzimas comercialmente disponíveis da classe oportuna que são suficientemente diferentes para não gerar erros durante a simulação da execução de uma máquina de Turing.
Finalmente, Rothemund mostra como seu método (que utiliza apenas reagentes comercialmente disponíveis e faz operações que são rotineiras para biólogos moleculares) pode ser aplicado para implementar uma Universal Turing Machine (UTM), já que Minsky projetou uma UTM com 7 estados e 4 símbolos (e 4 X 7 = 28 < 60). A expressão química de um modelo universal de computação melhora todas as demonstrações da capacidade computacional de um computador molecular hipotético.
O problema Busy Beaver para uma máquina de Turing com N estados (BB-N) consiste em projetar uma máquina de Turing com N estados e um alfabeto com 2 símbolos {Black, White} que escreve o maior número possível de símbolos Black antes de terminar (halt).